A head-to-head benchmarking of scRNA-seq CNV methods
GA, UNITED STATES, July 23, 2025 /EINPresswire.com/ -- Tumors are often genetic mosaics, harboring diverse cell populations shaped by mutations like copy number variations (CNVs). These genomic alterations drive cancer evolution—but detecting them at single-cell resolution remains challenging. In this study, scientists systematically evaluated five widely used tools that infer CNVs from single-cell RNA sequencing (scRNA-seq) data. The analysis uncovered dramatic differences in performance among methods, depending on data type and experimental design. Notably, CaSpER and CopyKAT emerged as top performers for CNV inference, while inferCNV and CopyKAT excelled in identifying tumor subpopulations. This comprehensive comparison provides a valuable roadmap for researchers aiming to decode cancer heterogeneity more precisely.
Cancer is not a single disease but a dynamic ecosystem of genetically distinct cells, each evolving under selective pressures. One of the key genomic mechanisms fueling this diversity is copy number variation (CNV)—where large DNA segments are duplicated or deleted—altering gene dosage and function. Although bulk sequencing can detect CNVs, it masks important single-cell differences. With the rise of scRNA-seq, researchers now have an opportunity to infer CNVs indirectly through transcriptomic signals. Yet, this task is far from straightforward. Inference methods vary widely in sensitivity, specificity, and robustness, and the lack of systematic evaluation has left researchers guessing which tool to trust. Due to these limitations, a rigorous benchmarking study was needed to guide accurate CNV detection using scRNA-seq.
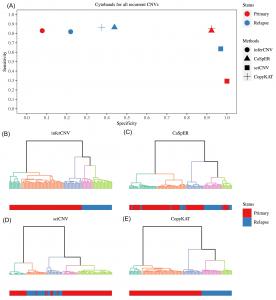
A multidisciplinary team led by researchers from Loma Linda University, the NCBI, and international partners addressed this knowledge gap with a large-scale benchmarking effort. Their findings were published on June 4, 2025, in Precision Clinical Medicine. The team rigorously tested five popular CNV inference methods—HoneyBADGER, inferCNV, sciCNV, CaSpER, and CopyKAT—across diverse scRNA-seq platforms and tumor models, including a newly generated clinical dataset from a small cell lung cancer patient. Their goal: to identify which tools deliver the most accurate and reliable CNV insights in real-world research settings.
The study applied each method to datasets from several distinct scRNA-seq platforms, paired tumor-normal cell lines, artificial mixtures of lung cancer cells, and clinical samples. Performance was measured using criteria such as sensitivity, specificity, and accuracy in identifying tumor subpopulations. CaSpER and CopyKAT consistently delivered the most balanced CNV inference results, though their effectiveness varied with sequencing depth and platform type. In contrast, inferCNV and sciCNV excelled in distinguishing tumor subclones when analyzing data from a single platform.
The team also tested each method's ability to detect rare tumor populations. InferCNV showed strong sensitivity—especially when enough cells were sequenced—while sciCNV and HoneyBADGER fell short in this regard. When combining datasets across platforms, batch effects severely impacted most methods, unless corrected using tools like ComBat. The allele-based version of HoneyBADGER, although less sensitive overall, proved more resilient to such batch-related distortions. Lastly, validation using clinical small cell lung cancer samples confirmed that CaSpER and CopyKAT yielded the most accurate CNV calls, while inferCNV and CopyKAT best identified relapsed subclones—underscoring the value of method-specific strengths.
"Understanding cancer at the single-cell level is essential for tackling tumor evolution and therapy resistance," said Prof. Charles Wang, co-corresponding author of the study. "Our benchmarking work provides the field with a clear reference point—highlighting not only which tools work best, but also under what conditions. It's a step toward more reliable and personalized cancer genomics."
This study offers a practical guide for selecting CNV inference tools tailored to specific scRNA-seq platforms and research goals. As single-cell genomics transitions from the bench to the clinic, the ability to accurately profile genetic alterations like CNVs is becoming critical for diagnostics, biomarker discovery, and tracking treatment response. These findings may also inform the next generation of algorithm development—aimed at overcoming current limitations such as platform bias and batch variability. Ultimately, better tools mean sharper insights into cancer's genetic landscape, paving the way for more targeted and effective therapies."
References
DOI
10.1093/pcmedi/pbaf011
Original Source URL
https://doi.org/10.1093/pcmedi/pbaf011
Funding information
The genomic work carried out at the LLU Center for Genomics was funded in part by the Ardmore Institute of Health grant 2150141 (CW) and Dr. Charles A. Sims' gift to LLU Center for Genomics.
Lucy Wang
BioDesign Research
email us here
Legal Disclaimer:
EIN Presswire provides this news content "as is" without warranty of any kind. We do not accept any responsibility or liability for the accuracy, content, images, videos, licenses, completeness, legality, or reliability of the information contained in this article. If you have any complaints or copyright issues related to this article, kindly contact the author above.
Bath Garden Center Offers Community Event for Families Offering Farm Animal Education for All Ages
Palms Up Leadership by Rich C. Plumery Redefines Leadership for the Modern Workplace
Lounge Lizard Worldwide Unveils 2025 Guide to Identifying and Preventing Black Hat SEO Threats
Więcej ważnych informacji
 Jedynka Newserii
Jedynka Newserii

 Jedynka Newserii
Jedynka Newserii
Prawo

Kolejne polskie miasta chcą być przyjazne dzieciom. Planują stworzyć najmłodszym dobre warunki do rozwoju
Cztery miasta w Polsce posiadają tytuł Miasta Przyjaznego Dzieciom nadany przez UNICEF Polska. Dziewięć kolejnych miast czeka na certyfikację, a w ostatnich miesiącach do programu zgłosiło się kilka następnych. Na całym świecie inicjatywa została przyjęta już w ponad 4 tys. samorządów, a w Hiszpanii objęła połowę dziecięcej populacji miast. Program UNICEF-u ma na celu zachęcenie włodarzy do traktowania najmłodszych obywateli w sposób podmiotowy, respektowania ich praw i zaproszenia ich do współdecydowania o przyszłości.
Przemysł
W ciągu roku w Polsce ubyło 500 przedsiębiorstw odzieżowo-tekstylnych. Problemem są spadki zamówień z Europy Zachodniej i wzrost kosztów

Wartość rynku odzieżowego w Polsce wynosi 66,9 mld zł, z czego 10 mld zł to wartość krajowej produkcji – wynika z danych PIOT. Od czasu pandemii branża mierzy się z szeregiem wyzwań, wśród których najpoważniejsze to wzrost kosztów pracy i produkcji, przerwane łańcuchy dostaw i spadek zamówień – zarówno w kraju, jak i za granicą, a także wzrost nieuczciwej konkurencji na rynku, czyli głównie importu z Chin. Skala wyzwań sprawia, że w ubiegłym roku z rynku zniknęło 500 firm. Producenci odzieży apelują do rządu o wsparcie.
Handel
D. Obajtek: Orlen powinien być o 30–40 proc. większą spółką. Byłoby to z korzyścią dla konsumentów

Orlen jest największym polskim przedsiębiorstwem. Jego przychody ze sprzedaży w 2024 roku wyniosły blisko 295 mln zł, a rok wcześniej – ponad 372 mln – wynika z raportu Rzeczpospolitej „Lista 500”. W ubiegłorocznym rankingu Fortune 500 uwzględniającym największe korporacje znalazł się na 216. miejscu na świecie i 44. w Europie. Według Daniela Obajtka, europosła PiS-u i byłego prezesa Orlenu, spółka powinna jeszcze urosnąć, tym samym gwarantując konsumentom szereg korzyści, a także przyspieszać inwestycje m.in. w obszarze petrochemii i energetyki zero- oraz niskoemisyjnej.
Partner serwisu
Szkolenia

Akademia Newserii
Akademia Newserii to projekt, w ramach którego najlepsi polscy dziennikarze biznesowi, giełdowi oraz lifestylowi, a także szkoleniowcy z wieloletnim doświadczeniem dzielą się swoją wiedzą nt. pracy z mediami.








.gif)

 |
| |
| |
|